IMPRONTA GENÓMICA Y REPRODUCCIÓN ASISTIDA
Cristina Camprubí
Grupo de Investigación Impronta genómica y Cáncer · Programa de Epigenética y Biología del Cáncer (PEBC)
Institut d’Investigació Biomèdica de Bellvitge (IDIBELL).
email:ccamprubi@idibell.cat
Resumen: La impronta genómica es una marca epigenética específica de alelo que regula la expresión de determinados genes del genoma en función de su origen parental, conllevando su expresión monoalélica. El término epigenética hace referencia a cambios heredables que no implican modificaciones en la secuencia del DNA. Estos cambios corresponden a la metilación del DNA y a modificaciones de las proteínas histonas. La impronta genómica se establece durante la gametogénesis y debe mantenerse después de la fecundación para el correcto desarrollo embrionario. En los últimos años, numerosos trabajos han correlacionado anomalías en la impronta genómica con la aplicación de técnicas de reproducción asistida o con la infertilidad. Profundizar en este área de conocimiento contribuirá en mejorar tanto el éxito como la seguridad de estas técnicas. Rev Asoc Est Biol Rep 2010; 15(1):36-41.
Palabras clave: Impronta genómica, Epigenética, Reproducción asistida
IMPRINTING AND ASSISTED REPRODUCTION
Abstract: Imprinting is an allelic-specific epigenetic mark that controls certain genes expression on the basis of the parental origin resulting in monoallelic expression. Epigenetics reffers to heritable changes that do not involve modifications in the DNA sequence. These changes correspond to DNA methylation and histone modifications. Imprinting is established during gametogenesis and it must be maintained after fertilization for proper embryo development. In the last years several reports correlate imprinting abnormalities with assisted reproduction techniques or with infertility itself. The increase of knowledge of this field will contribute to improve the success and safety of these technologies. Rev Asoc Est Biol Rep 2010; 15(1):36-41.
Key words: Imprinting, Epigenetics, Assisted reproduction
Desde el nacimiento de Louise Brown a finales de los años 70, el desarrollo y aplicación de técnicas de reproducción asistida (TRA) ha permitido superar muchos casos de infertilidad. Sin embargo, considerando su impacto social, cabe plantearse si la investigación básica asociada a las TRA avanza de forma sincrónica a su aplicación, con el objetivo de incrementar el conocimiento de la biología de gametos y embriones procedentes de la población infértil, aportando así información de gran relevancia que permita, tanto mejorar el éxito del ciclo de reproducción, como garantizar la seguridad para la descendencia.
En el área de la genética humana se ha observado un incremento de individuos portadores de anomalías cromosómicas constitucionales (de Braekeleer and Dao, 1991), de anomalías meióticas (Egozcue et al., 1983) o de gametos cromosómicamente desequilibrados (Arán et al., 1999; Vegetti et al., 2000; Rubio et al., 2001) en poblaciones de individuos con problemas de fertilidad. El estudio del cariotipo de la pareja, el diagnóstico genético preimplantacional y estudios citogenéticos en células germinales, permiten ofrecer consejo genético y acotar el riesgo de que la descendencia pueda heredar anomalías cromosómicas o genéticas, en el caso de que alguno de los progenitores sea portador de anomalías cromosómicas o monogénicas, en casos de abortos de repetición o edad avanzada, o en casos en los que existe un bloqueo meiótico causado por un incremento de anomalías cromosómicas en las células germinales.
Mientras que los riesgos genéticos cada vez están más acotados, en la actualidad la investigación también se centra en elucidar si existen riesgos epigenéticos asociados a la aplicación de TRA.
Epigenética e impronta genómica.
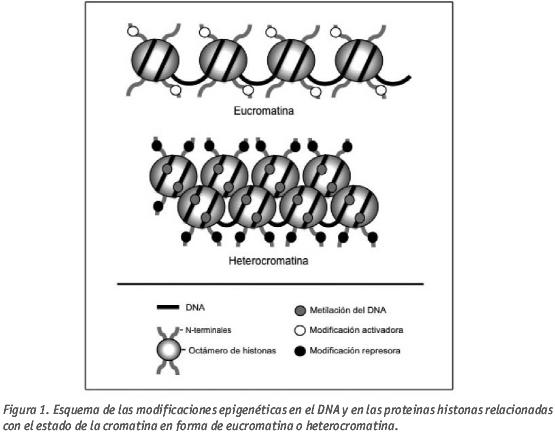
El término epigenética se refiere a cambios heredables que no implican variaciones en la secuencia de nucleótidos en la molécula de DNA, pero sí regulan la expresión génica. Estos cambios incluyen la metilación del DNA en el carbono 5 de las bases citosina en dinucleótidos CpG y modificaciones postraduccionales en los extremos N- terminales de las histonas. Estas modificaciones covalentes actúan conjuntamente modificando la conformación de la cromatina. El DNA de células eucariotas se encuentra empaquetado mediante la formación de nucleosomas que consisten en 146 pares de bases de la molécula de DNA, organizadas alrededor de un octámero de 4 proteinas histonas (H2A, H2B, H3 y H4). Dependiendo del estado de metilación del DNA, así como de modificaciones represoras o activadoras de las proteínas histonas (principalmente metilaciones y acetilaciones en aminoácidos lisina), la cromatina adopta una conformación cerrada (heterocromatina) o abierta (eucromatina) (Figura 1). La conformación de la cromatina en forma de heterocromatina se asocia a represión génica, dado que el DNA es inaccesible a la maquinaria de transcripción, mientras que la conformación en forma de eucromatina se asocia a expresión génica.
El término epigenética engloba la impronta genómica, un sistema de regulación de la expresión génica, presente en especies de gestación intrauterina (Das et al., 2009), que consiste en marcar diferencialmente, mediante modificaciones epigenéticas, determinados genes en función de su origen parental, de manera que sólo se expresa el alelo materno o paterno. Considerando el comportamiento diferencial de alelo de estos genes, la impronta genómica es, junto con los caracteres ligados a los cromosomas sexuales y a los genes mitocondriales, una de las excepciones al principio mendeliano básico de equivalencia, el cual describe la inexistencia de diferencias en el comportamiento de los genes de origen paterno y materno.
Las primeras evidencias de la existencia de impronta genómica surgieron de los resultados obtenidos en experimentos de transferencia nuclear. En 1984 Surani y colaboradores y McGrath y Solter crearon embriones murinos ginogenéticos y androgenéticos, es decir, embriones con dos pronúcleos o genomas maternos o con dos pronúcleos o genomas paternos, respectivamente (Surani et al., 1984; McGrath and Solter, 1984). La presencia del genoma materno y paterno implica el desarrollo de un embrión y tejidos trofoblásticos normales. En cambio, los embriones ginogenéticos crecen relativamente bien, pero los tejidos trofoblásticos (la placenta) son atróficos. En el caso de los androgenéticos, los embriones apenas se desarrollan aunque las placentas son de gran tamaño (Surani et al., 1984; McGrath and Solter, 1984). La conclusión inmediata frente a estos resultados es que la contribución genética paterna es primordial para el desarrollo de la placenta y la materna lo es para el correcto desarrollo embrionario.
Otra evidencia de la existencia de impronta genómica proviene del hecho de que disomías uniparentales (DUP) de ciertos cromosomas tengan como consecuencia el desarrollo de síndromes o anomalías del crecimiento. Este es el caso de los síndromes de Prader-Willi y Angelman, que pueden ser originados por DUP materna o paterna del cromosoma 15, respectivamente (Cassidy and Schwartz, 1998; Cassidy et al., 2000) y de los síndromes de Beckwith-Wiedemann y de Silver- Russell, que pueden ser causados por DUP paterna del cromosoma 11 y DUP materna del cromosoma 7, respectivamente (Eggermann et al.,2008). Es también ampliamente conocido que DUP del cromosoma 14 causa diferentes fenotipos de crecimiento anómalo, dependiendo del origen de la DUP (Murphy et al., 2003).
En resumen, determinadas anomalías fenotípicas pueden ser causadas por la herencia de dos copias maternas o paternas de determinados genes, manifestando la presencia de genes con un comportamiento específico de alelo y su relevancia en el desarrollo. Además de DUP, anomalías epigenéticas que conllevan la expresión bialélica (ausencia de metilación en ambas copias) o no expresión (presencia de metilación en ambas copias) de genes regulados por impronta genómica, pueden resultar no sólo en el desarrollo de síndromes y anomalías del crecimiento, sino también en el desarrollo de cáncer, diabetes neonatal transitoria (Temple, 2007) y pseudohipoparatiroidismo (Bastepe, 2008), dependiendo del gen o genes afectados. Considerando la función de determinados genes regulados por impronta genómica en el desarrollo cerebral y fetal, la etiología del autismo y de la esquizofrenia (Badcock and Crespi, 2006) así como la preeclampsia (Enquobahrie et al., 2008), la restricción del crecimiento intrauterino (McMinn et al., 2006; Diplas et al., 2009) y el bajo desarrollo para la edad gestacional (Guo et al., 2008) han sido relacionados con anomalías en la impronta genómica.
Actualmente se conocen aproximadamente 60 genes regulados por impronta genómica en humanos, mapados en diversos cromosomas. Cabe comentar que los genes regulados por impronta genómica suelen encontrarse agrupados en dominios cromosómicos formando un cluster de genes. La expresión monoalélica de todos los genes agrupados en un determinado dominio, se encuentra bajo el control de una única región, denominada centro regulador de la impronta (Imprinting Control Region; ICR). Las ICR del genoma se corresponden con lo que se conoce como región diferencialmente metilada (Differentially DNA-Methylated Region; DMR). Estas regiones del genoma contienen islas CpG (regiones con un alto contenido de CpG) y son en las que se establece la metilación del DNA y las modificaciones represoras de histonas que conllevan la heterocromatinización de toda la región de forma específica de alelo.
Herencia epigenética y de la impronta genómica
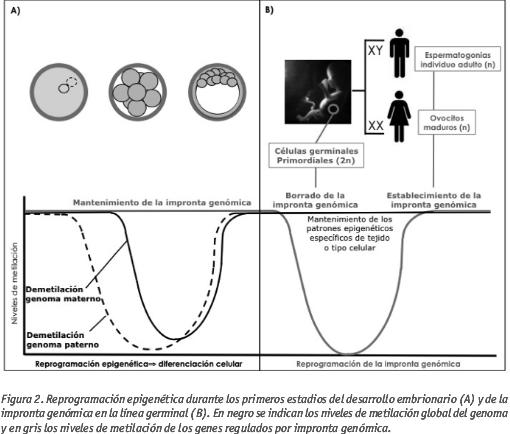
Después de la fecundación, el zigoto resultante hereda la copia materna y paterna de todos los genes del genoma, los cuales deben transmitirse de forma fiel a lo largo de la posterior proliferación celular, asegurando que todas las células del organismo compartan la misma información genética. Para el desarrollo es también imprescindible la diferenciación celular. Del hecho de que células genéticamente idénticas sean tan diferentes funcional y morfológicamente, es responsable la epigenética, la cual regula la expresión o represión de unos u otros genes del genoma en función del tipo celular. Por ello, después de la fecundación y durante las primeras etapas del desarrollo embrionario, los patrones epigenéticos específicos de ovocito y espermatozoide son eliminados mediante una desmetilación global de ambos genomas. En el estadio de blastocisto, emergen los patrones epigenéticos específicos de cada tipo celular (Figura 2A). Estos patrones deben ser transmitidos a través de la mitosis, para conservar el estado de diferenciación y funcionalidad en las células hijas.
A pesar de la flexibilidad epigenética, que permite la diferenciación celular, los patrones epigenéticos que controlan la expresión monoalélica de genes regulados por impronta genómica son, en términos generales, comunes en todos los tipos celulares, y es especialmente crucial que se mantengan desde la fecundación y a lo largo de las primeras etapas de la embriogénesis, para el correcto desarrollo del embrión, de los tejidos extraembrionarios y para el correcto desarrollo neurológico (Figura 2A). Por lo tanto, la metilación diferencial de alelo de los genes regulados por impronta genómica debe ser heredada de ovocitos y espermatozoides, y debe mantenerse o protegerse frente a la desmetilación global que permite la reprogramación epigenética del genoma. Para asegurar la transmisión de la metilación monoalélica de estos genes, existe el mecanismo de reprogramación de la impronta genómica en la línea germinal (Figura 2B). Este ciclo de reprogramación evita que el 50% de los gametos haploides, procedentes de las células germinales primordiales diploides, sean portadores de la copia metilada y el otro 50% sean portadores de la no metilada. Frente a esta situación, un ovocito portador de la copia metilada de un determinado gen regulado por impronta genómica, podría ser fecundado por un espermatozoide también portador de la copia metilada, de modo que el zigoto resultante sería portador de una anomalía en la impronta genómica dado que ambas copias del gen se encontrarían inactivas o silenciadas, conllevando a posibles anomalías del desarrollo. Del mismo modo, si un ovocito portador de la copia no metilada fuera fecundado por un espermatozoide también portador de la copia no metilada, el zigoto resultante expresaría ambas copias del gen, hecho que también se correlaciona con una anomalía en la impronta genómica, con consecuencias fenotípicas. Para evitar esta situación, en las células germinales primordiales la impronta genómica se borra, y se establecen los patrones de metilación específicos de alelo de los genes regulados por impronta genómica durante la gametogénesis, en función del sexo. Así, aquellos genes en los que su patrón de impronta se correlaciona con metilación materna, establecerán dicha metilación durante la ovogénesis en todos los ovocitos. El establecimiento de la impronta materna se realiza durante la maduración ovocitaria (Sato et al., 2007), habiéndose considerado por algunos autores que ésta no se completa hasta el momento de la fecundación o justo después de ésta (El- Maarri et al., 2001). En el caso de los genes en los que su patrón de impronta se correlaciona con metilación paterna, ésta se establecerá durante la espermatogénesis, siendo completa en las espermatogonias del individuo adulto (Kerjean et al., 2000). De este modo, todos los ovocitos y espermatozoides serán portadores de la impronta específica de alelo para cada uno de los genes regulados por impronta genómica, asegurando la herencia de un patrón normal de metilación de una de las dos copias de estos genes, y, por ende, asegurando su posterior expresión monoalélica durante el desarrollo.
Impronta genómica y Técnicas de reproducción asistida
Tanto la metilación del DNA como las modificaciones en las proteínas histonas son susceptibles de presentar alteraciones que pueden ser consecuencia de: factores genéticos (anomalías en los genes que codifican factores implicados en el establecimiento y/o mantenimiento de la metilación) (Zetterberg et al., 2003; Park et al., 2005; Lee et al., 2006; Zogel et al., 2006; Kobayashi et al., 2009); factores ambientales intrauterinos (Lim et al., 2010); exposición a toxinas (Anway and Skinner, 2008); tratamientos hormonales (Pathak et al., 2008, 2010); diferencias dietéticas (Chmurzynska, 2010).
El conocimiento sobre los mecanismos y las etapas de establecimiento de la impronta genómica, junto con la aparición de datos epidemiológicos que relacionan un moderado incremento en el riesgo del desarrollo de síndromes relacionados con anomalías en la impronta genómica en la población de niños concebidos mediante reproducción asistida (Cox et al., 2002; Ørstavik et al., 2003; Maher et al., 2003; Gicquel et al., 2003; DeBaun et al., 2003), ha focalizado la atención de especialistas en epigenética e impronta genómica en la investigación relacionada con la reproducción asistida. En todos los casos descritos en estos estudios epidemiológicos, la causa del síndrome es la pérdida de metilación (Loss of Imprinting -LOI-) en el alelo materno.
Cultivo in vitro de embriones, hiperestimulación ovárica y maduración in vitro de ovocitos
La aplicación de TRA implica mayoritariamente la fecundación y el desarrollo embrionario temprano en condiciones in vitro, siendo omitido del proceso el ambiente uterino. Existen múltiples datos experimentales obtenidos de modelos animales, que avalan una relación entre el cultivo in vitro de embriones, así como la composición de los medios de cultivo y la transferencia embrionaria en condiciones de asincronía con el estado uterino, y el desarrollo del síndrome de sobrecrecimiento, conocido como Large offspring syndrome (LOS) y causado por anomalías en genes regulados por impronta genómica (Young et al., 1998; Sinclair et al., 2000; Young et al., 2001; Mann et al., 2004; Li et al., 2005). Además de los efectos fenotípicos inmediatos, debe considerarse la posibilidad de que el cultivo in vitro pueda tener efectos a largo plazo. En este contexto, se ha descrito una relación entre el desarrollo de obesidad, ansiedad y déficit de memoria asociados a anomalías en la impronta, en descendencia obtenida mediante cultivo in vitro en el modelo murino (Fernández-Gonzalez et al., 2007). Cabe mencionar que en embriones murinos obtenidos por transferencia intrauterina de zigotos obtenidos in vivo, también se han descrito LOI, concretamente en tejidos extraembrionarios. Estas anomalías se intensifican y están presentes, no solo en los tejidos extraembrionarios sino también en el embrión, cuando los embriones son cultivados in vitro antes de su transferencia (Rivera et al., 2008). Este hecho plantea que, así como la manipulación de embriones previa a su implantación puede tener un efecto deletéreo sobre el mantenimiento de la impronta genómica per se, la práctica metodológica común en ambos casos es la hiperestimulación ovárica.
En la práctica habitual de técnicas de reproducción asistida, es complicado aislar la exposición a hormonas, que a su vez conlleva la maduración de múltiples ovocitos inducida por el tratamiento hormonal, y el posterior cultivo embrionario.
Considerando que el período de establecimiento de la impronta genómica materna es coincidente con la maduración de los ovocitos, es lógico plantearse que tanto la hiperestimulación ovárica como la maduración in vitro de ovocitos también puedan influir en la impronta, en este caso en su establecimiento. En el año 2007, Sato y colaboradores presentaron un estudio de metilación de diversos genes regulados por impronta genómica, realizado en ovocitos de ratón y humanos en diferentes estados de maduración y en ovocitos madurados in vitro. En el caso de los ovocitos humanos, los autores analizaron tanto ovocitos inmaduros procedentes de exámenes laparoscópicos de población, así como ovocitos en diferentes estados de maduración obtenidos mediante hiperestimulación de pacientes infértiles (Sato et al., 2007). Sus resultados apuntan de forma consistente la presencia de LOI de diversos genes regulados por impronta, que correlacionan tanto con la hiperestimulación ovárica como con la maduración in vitro de ovocitos. Además, en un estudio reciente del efecto de la hiperestimulación en la impronta genómica, realizado en modelo murino, se ha descrito un efecto dosis-dependiente sobre la metilación del DNA, no sólo en forma de pérdida de metilación sino también por la presencia de metilación aberrante en el alelo materno en genes en los que la metilación se establece en el paterno (Market-Velker et al., 2010). A pesar de ser resultados concluyentes y que en modelo murino queda excluido el componente infértil, éste no debe ser excluido en humanos como consecuencia de la presencia de anomalías en la impronta genómica.
Infertilidad y anomalías en la impronta genómica
Del mismo modo en que anomalías genéticas o cromosómicas explican casos de bloqueo meiótico, anomalías en la impronta genómica pueden explicar casos de infertilidad idiopática. Este planteamiento no puede ser demostrado experimentalmente en el caso de la meiosis femenina, por la limitación de obtener ovocitos sin hiperestimulación. Aún así, a partir de datos epidemiológicos obtenidos de madres de pacientes con síndrome de Angelman, causado por ausencia de metilación materna, Ludwig y colaboradores demostraron una correlación entre infertilidad y la presencia de estas anomalías (Ludwig et al., 2005). En el caso de la meiosis masculina, han sido publicados múltiples trabajos experimentales confirmando la relación entre la presencia de anomalías en la impronta y oligo-, terato- y astenozoospermia (Marques et al., 2004; Kobayashi et al., 2007; Marques et al., 2008, 2009; Kobayashi et al., 2009; Boissonnas et al., 2009; Poplinsky et al., 2009; Hammoud et al., 2009). Cabe apuntar que, a pesar de que los resultados de estos trabajos avalan con claridad la presencia de anomalías en la impronta e infertilidad, en la mayoría de ellos no se incluyen poblaciones control de referencia, por lo que dicha relación podría tener una menor incidencia que la descrita hasta la actualidad en la literatura. Si la presencia de este tipo de anomalías en espermatozoides de individuos infértiles fuera tan elevada como la sugerida en estos trabajos, cabría esperar una mayor incidencia de síndromes causados por anomalías en la impronta paterna en la descendencia concebida mediante técnicas de reproducción asistida. Contrariamente, no existen datos epidemiológicos que apunten a un incremento en la incidencia de estos síndromes causados por anomalías paternas, a diferencia de los datos que sí apuntan a un incremento moderado de síndromes causados por anomalías maternas. Sin embargo, y teniendo en cuenta que los genes paternos contribuyen mayoritariamente al desarrollo de tejidos extraembrionarios, resulta atractivo plantear la hipótesis de que las anomalías presentes en espermatozoides de individuos infértiles puedan ser, o bien más deletéreas para la implantación y/o gestación, o bien puedan quedar confinadas a la placenta y verse reflejadas en una mayor incidencia de restricción del crecimiento uterino, consecuencia de una transferencia limitada o anómala de nutrientes de la madre al feto. Será interesante aportar datos experimentales que contribuyan a confirmar o rehusar esta hipótesis.
Referencias
Anway MD, Skinner MK. Epigenetic programming of the germ line: effects of endocrine disruptors on the development of transgenerational disease. Reprod Biomed Online 2008; 16(1):23-25
Aran B, Blanco J, Vidal F, Vendrell JM, Egozcue S, Barri PN, et al. Screening for abnormalities of chromosomes X, Y, and 18 and for diploidy in spermatozoa from infertile men participating in an in vitro fertilization- intracytoplasmic sperm injection program. Fertil Steril 1999; 72:696-701
Badcock C, Crespi B. Imbalanced genomic imprinting in brain development: an evolutionary basis for the aetiology of autism. J Evol Biol 2006; 19:1007–1032
Bastepe M. The GNAS locus and pseudohypoparathyroidism. Adv Exp Med Biol 2008; 626:27-40.
Boissonnas CC, Abdalaoui HE, Haelewyn V, Fauque P, Dupont JM, Gut I, et al. Specific epigenetic alterations of IGF2-H19 locus in spermatozoa from infertile men. Eur J Hum Genet 2010; 18(1):73-80.
Cassidy SB, Schwartz S. Prader-Willi and Angelman syndromes. Disorders of genomic imprinting. Medicine 1998; 77:140-151
Cassidy SB, Dykens E, Williams CA. Prader- Willi and Angelman syndromes: sister imprinted disorders. Am J Med Genet 2000; 97(2):136-146.
Chmurzynska A. Fetal programming: link between early nutrition, DNA methylation, and complex diseases. Nutr Rev 2010; 68(2):87-98
Cox GF, Bürger J, Lip V, Mau UA, Sperling K, Wu BL, et al. Intracytoplasmic sperm injection may increase the risk of imprinting defects. Am J Hum Genet 2002; 71(1):162-164
Das R, Hampton DD, Jirtle RL. Imprinting evolution and human health. Mamm Genome. 2009; 20(9-10):563-572
DeBaun MR, Niemitz EL, Feinberg AP. Association of in vitro fertilization with Beckwith-Wiedemann syndrome and epigenetic alterations of LIT1 and H19. Am J Hum Genet. 2003 Jan;72(1):156-160
De Braekeleer and Dao. Cytogenetic studies in male infertility: a review. Hum Reprod 1991; 6:245-250
Diplas AI, Lambertini L, Lee MJ, Sperling R, Lee YL, Wetmur J et al. Differential expression of imprinted genes in normal and IUGR human placentas. Epigenetics 2009; 4(4):235-240.
Eggermann T, Eggermann K, Schönherr N. Growth retardation versus overgrowth: Silver- Russell syndrome is genetically opposite to Beckwith-Wiedemann syndrome. Trends Genet 2008; 24(4):195-204
Egozcue J, Templado C, Vidal F, Navarro J, Morer-Fargas F, Marina S. Meiotic studies in a series of 1100 infertile and sterile males. Hum Genet 1983; 65:185-188.
El-Maarri O, Buiting K, Peery EG, Kroisel PM, Balaban B, Wagner K, et al. Maternal methylation imprints on human chromosome 15 are established during or after fertilization. Nat Genet 2001; 27(3):341-344.
Enquobahrie DA, Meller M, Rice K, Psaty BM, Siscovick DS, Williams MA. Differential placental gene expression in preeclampsia. Am J Obstet Gynecol 2008; 199(5):566.e1-11
Fernández-Gonzalez R, Ramirez MA, Bilbao A, De Fonseca FR, Gutiérrez-Adán A. Suboptimal in vitro culture conditions: an epigenetic origin of long-term health effects. Mol Reprod Dev. 2007 Sep;74(9):1149-1156
Gicquel C, Gaston V, Mandelbaum J, Siffroi JP, Flahault A, Le Bouc Y. In vitro fertilization may increase the risk of Beckwith-Wiedemann syndrome related to the abnormal imprinting of the KCN1OT gene. Am J Hum Genet. 2003; 72(5):1338-1341
Guo L, Choufani S, Ferreira J, Smith A, Chitayat D, Shuman C et al. Altered gene expression and methylation of the human chromosome 11 imprinted region in small for gestational age (SGA) placentae. Dev Biol 2008; 320(1):79-91
Hammoud SS, Purwar J, Pflueger C, Cairns BR, Carrell DT. Alterations in sperm DNA methylation patterns at imprinted loci in two classes of infertility. Fertil Steril. 2009 Oct 30. [Epub ahead of print]
Kerjean A, Dupont JM, Vasseur C, Le Tessier D, Cuisset L, Pàldi A, et al. Establishment of the paternal methylation imprint of the human H19 and MEST/PEG1 genes during spermatogenesis. Hum Mol Genet 2000; 9(14):2183-2187
Kobayashi H, Sato A, Otsu E, Hiura H, Tomatsu C, Utsunomiya T, et al. Aberrant DNA methylation of imprinted loci in sperm from oligospermic patients. Hum Mol Genet 2007; 16(21):2542-2551
Kobayashi H, Hiura H, John RM, Sato A, Otsu E, Kobayashi N, et al. DNA methylation errors at imprinted loci after assisted conception originate in the parental sperm. Eur J Hum Genet 2009; 17(12):1582-1591.
Lee HC, Jeong YM, Lee SH, Cha KY, Song SH, Kim NK, et al. Association study of four polymorphisms in three folate-related enzyme genes with non-obstructive male infertility. Hum Reprod 2006; 21(12):3162-3170
Li T, Vu TH, Ulaner GA, Littman E, Ling JQ, Chen HL, et al. IVF results in de novo DNA methylation and histone methylation at an Igf2-H19 imprinting epigenetic switch. Mol Hum Reprod 2005; 11(9):631-640.
Lim AL, Ferguson-Smith AC. Genomic imprinting effects in a compromised in utero environment: implications for a healthy pregnancy. Semin Cell Dev Biol 2010; 21(2):201-208
Ludwig M, Katalinic A, Gross S, Sutcliffe A, Varon R, Horsthemke B. Increased prevalence of imprinting defects in patients with Angelman syndrome born to subfertile couples. J Med Genet 2005; 42(4):289-291
Maher ER, Brueton LA, Bowdin SC, Luharia A, Cooper W, Cole TR, et al. Beckwith- Wiedemann syndrome and assisted reproduction technology (ART). J Med Genet 2003; 40(1):62-64.
Mann MR, Lee SS, Doherty AS, Verona RI, Nolen LD, Schultz RM, et al. Selective loss of imprinting in the placenta following preimplantation development in culture. Development 2004; 131(15):3727-3735
Market-Velker BA, Zhang L, Magri LS, Bonvissuto AC, Mann MR. Dual effects of superovulation: loss of maternal and paternal imprinted methylation in a dose- dependent manner. Hum Mol Genet 2010; 19(1):36-51
Marques CJ, Carvalho F, Sousa M, Barros A. Genomic imprinting in disruptive spermatogenesis. Lancet 2004;363(9422):1700-1702.
Marques CJ, Costa P, Vaz B, Carvalho F, Fernandes S, Barros A, et al. Abnormal methylation of imprinted genes in human sperm is associated with oligozoospermia. Mol Hum Reprod 2008; 14(2):67-7
Marques CJ, Francisco T, Sousa S, Carvalho F, Barros A, Sousa M. Methylation defects of imprinted genes in human testicular spermatozoa. Fertil Steril 2009 Apr 1. [Epubahead of print]
McGrath J, Solter D. Completion of mouse embryogenesis requires both the maternal and paternal genomes. Cell 1984 37(1):179-183
McMinn J, Wei M, Schupf N, Cusmai J, Johnson EB, Smith AC et al. Unbalanced placental expression of imprinted genes in human intrauterine growth restriction. Placenta 2006; 27(6-7):540-549
Murphy SK, Wylie AA, Coveler KJ, Cotter PD, Papenhausen PR, Sutton VR, et al. Epigenetic detection of human chromosome 14 uniparental disomy. Hum Mutat 2003; 22(1):92-97
Ørstavik KH, Eiklid K, van der Hagen CB, Spetalen S, Kierulf K, Skjeldal O, et al. Another case of imprinting defect in a girl with Angelman syndrome who was conceived by intracytoplasmic semen injection. Am J Hum Genet 2003; 72(1):218-219
Park JH, Lee HC, Jeong YM, Chung TG, Kim HJ, Kim NK, et al. MTHFR C677T polymorphism associates with unexplained infertile male factors. J Assist Reprod Genet 2005; 22(9-10):361-368
Pathak S, Kedia-Mokashi N, Saxena M, D’Souza R, Maitra A, Parte P et al. Effect of tamoxifen treatment on global and insulin-like growth factor 2-H19 locus-specific DNA methylation in rat spermatozoa and its association with embryo loss. Fertil Steril 2009; 91(5 Suppl):2253-2263
Pathak S, D’Souza R, Ankolkar M, Gaonkar R, Balasinor NH. Potential role of estrogen in regulation of the insulin-like growth factor2-H19 locus in the rat testis. Mol Cell Endocrinol 2010; 314(1):110-117
Poplinski A, Tüttelmann F, Kanber D, Horsthemke B, Gromoll J. Idiopathic male infertility is strongly associated with aberrant methylation of MEST and IGF2/H19 ICR1. Int J Androl. 2009 Oct 30. [Epub ahead of print]
Rivera RM, Stein P, Weaver JR, Mager J, Schultz RM, Bartolomei MS. Manipulations of mouse embryos prior to implantation result in aberrant expression of imprinted genes on day 9.5 of development. Hum Mol Genet 2008; 17(1):1-14.
Rubio C, Gil-Salom M, Simón C, Vidal F, Rodrigo L, Mínguez Y, et al. Incidence of sperm chromosomal abnormalities in a risk population: relationship with sperm quality and ICSI outcome. Hum Reprod 2001; 16:2084-2092
Sato A, Otsu E, Negishi H, Utsunomiya T, Arima T. Aberrant DNA methylation of imprinted loci in superovulated oocytes. Hum Reprod 2007; 22(1):26-35.
Sinclair KD, Young LE, Wilmut I, McEvoy TG. In-utero overgrowth in ruminants following embryo culture: lessons from mice and a warning to men. Hum Reprod 2000; 15 Suppl 5:68-86
Surani, M. A., S. C. Barton and M. L. Norris. Development of reconstituted mouse eggs suggests imprinting of the genome during gametogenesis. Nature 1984; 308:548–550
Temple IK. Imprinting in human disease with special reference to transient neonatal diabetes and Beckwith-Wiedemann syndrome. Endocr Dev 2007; 12:113-123.
Vegetti W, Van Assche E, Frias A, Verheyen G, Bianchi MM, Bonduelle M, et al. Correlation between semen parameters and sperm aneuploidy rates investigated by fluorescence in-situ hybridization in infertile men. Hum Reprod 2000; 15:351- 365
Young LE, Sinclair KD, Wilmut I. Large offspring syndrome in cattle and sheep. Rev Reprod 1998; 3(3):155-163
Young LE, Fernandes K, McEvoy TG, Butterwith SC, Gutierrez CG, Carolan C et al. Epigenetic change in IGF2R is associated with fetal overgrowth after sheep embryo culture. Nat Genet 2001; 27(2):153-154
Zetterberg H, Zafiropoulos A, Spandidos DA, Rymo L, Blennow K. Gene-gene interaction between fetal MTHFR 677C>T and transcobalamin 776C>G polymorphisms in human spontaneous abortion. Hum Reprod 2003; 18(9):1948-1950
Zogel C, Böhringer S, Gross S, Varon R, Buiting K, Horsthemke B. Identification of cis- and trans-acting factors possibly
modifying the risk of epimutations on chromosome 15. Eur J Hum Genet 2006; 14(6):752-758.