METABOLÓMICA Y REPRODUCCIÓN HUMANA
Imma Sánchez Ribas1,2, Francisco Domínguez2, Carlos Simon3.
1IVI Barcelona, Fundación IVI, Embryomics
2Embryomics
3IVI Valencia, Fundación IVI
INTRODUCCIÓN
Uno de los mayores retos en el campo de la reproducción humana es la selección de los embriones más apropiados a transferir. Hasta la fecha, en los ciclos de Fecundación In Vitro, la evaluación morfológica sigue siendo el método de asesoramiento utilizado para escoger el mejor embrión o blastocisto, pero este método de selección es altamente subjetivo y su modesto poder predictivo y la variabilidad inter- e intra- observador limita su valor.
Además, seleccionar el mejor embrión a transferir no sólo significa escoger el embrión que nos va a dar más posibilidades de embarazo, sino que también es crucial escoger aquel embrión que nos va a aumentar la tasa de recién nacido vivo en casa.
Actualmente, lo más novedoso en el estudio de la embriogénesis son las técnicas -Ómicas. Son disciplinas que estudian los eventos e interacciones de las estructuras celulares y sus procesos, desde el DNA hasta la función biológica; es decir, desde los genes hasta los metabolitos. Todos los procesos que contribuyen en el fenotipo pasan a través de este complejo sistema, y la Genómica, Transcriptómica, Proteómica y Metabolómica intentan comprender cómo un embrión crece y cuáles son sus indicadores de éxito.
Las tecnologías proteómica y metabolómica han desarrollado el análisis cualitativo y cuantitativo de proteínas y metabolitos, respectivamente, en una célula, un organismo o una muestra bajo distintas condiciones, con la finalidad de obtener un perfil comprensible de todas las proteínas y metabolitos de esa célula, organismo o muestra. Los metabolitos de bajo peso molecular representan el producto final de procesos de regulación celular, y por lo tanto revela la respuesta de los sistemas biológicos a una variedad de influencias genéticas, nutricionales y ambientales (Singh and Sinclair, 2007).
Aunque la metabolómica es obviamente complementaria a la genómica y proteómica, tiene ventajas especiales. La tabla número 1 recoge las ventajas del estudio del metaboloma en comparación con el del transcriptoma y el proteoma (Dunn and Ellis, 2003).
CAMBIO EN LAS ESTRATEGIAS DE GENERACION DE HIPÓTESIS
Las -Ómicas están transformando el método de investigación. El obtener tal grado de información ha implicado un cambio en el ciclo del conocimiento. En el ciclo del conocimiento tradicional, el conocimiento previo se utilizaba para construir una hipótesis para ser testada experimentalmente. El experimento producía datos que daban consistencia o no a la hipótesis inicial. Es decir, la hipótesis era el punto de partida. El acercamiento, por tanto, era hipotético- deductivo.
Sin embargo, con las -Ómicas tenemos una situación de acercamiento inductivo, donde no hay una hipótesis real, y la estrategia es generar esta hipótesis desde los datos obtenidos, provocando un descubrimiento del conocimiento y pudiendo a posteriori testar estas nuevas hipótesis de la forma tradicional. El diseño experimental es muy importante en el acercamiento inductivo, porque determinará qué experimentos nos van a dar la información que estamos buscando. La estrategia en la que un algoritmo elige qué experimentos hacer es conocida como “aprendizaje activo” y es la estrategia de elección. Para ello debemos contar con sistemas informáticos de alta complejidad (Waidyanathan, 2003; Goodacre, 2004)
¿QUÉ ES LA METABOLÓMICA?
Existe un debate activo en la comunidad científica sobre la definición exacta de metaboloma. Fue definido por primera vez por Oliver y cols. (1988) como el complemento cuantitativo de todas las moléculas de bajo peso molecular que se encuentran en las células en un estado fisiológico particular o de desarrollo. Otra definición sustenta que el metaboloma consiste en sólo aquellas moléculas pequeñas y nativas que están participando en reacciones metabólicas generales y que son requeridas para el mantenimiento, crecimiento y funcionamiento de la célula (Beecher et al., 2003).
En términos generales, por metabolómica (Nicholson et al., 1999; Fiehn et al., 2002) se entiende la obtención, de una manera simultánea y global, de perfiles correspondientes a múltiples concentraciones de metabolitos y de sus fluctuaciones sistémicas y celulares en respuesta a fármacos, dieta, genética, estilo de vida, entorno y estímulos, de forma que sea posible caracterizar los efectos adversos y beneficiosos de esas interacciones.
La metabolómica busca una descripción analítica de muestras biológicas complejas, e intenta caracterizar y cuantificar todas las moléculas pequeñas de ese tipo de muestras. Sin embargo, medir el metaboloma es un reto analítico considerable. Esto es debido a la naturaleza lábil de los metabolitos, el rango ampliamente dinámico y diverso de muchos de ellos, la complejidad química y heterogénea de los mismos, la falta de técnicas analíticas automatizadas que puedan medir cuantitativamente un gran número de metabolitos de estructura desconocida, el rendimiento de las mediciones y la naturaleza elaborada de los protocolos de extracción (Whitfield et al., 2004).
PLATAFORMAS DE ANÁLISIS
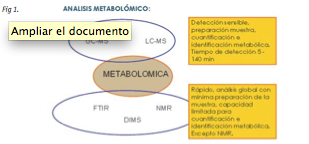
Todos estos desafíos deben ser abordados por la plataforma de análisis empleados. Las plataformas para las medidas del metaboloma deben ser imparciales, sensibles y con una gran capacidad de análisis de alto rendimiento para discriminar un gran número de metabolitos. A pesar del gran éxito en el desarrollo de la tecnología metabolómica, aún no es posible analizar con una única plataforma analítica la diversidad de metabolitos complejos. La Fig. 1 indica las distintas tecnologías para el análisis metabolómico de muestras, así como las características más relevantes de las mismas. El tipo de muestras que se analiza mediante estas técnicas son biofluidos como orina, plasma o suero previa precipitación de proteínas; metabolitos intracelulares de plantas, microbios y animales, previa extracción mediante solventes polares y no polares; tejidos sin previa o mínima preparación y huellas metabólicas en secreciones naturales de metabolitos intracelulares hacia el volumen extracelular.
De todas ellas, las tecnologías más empleadas para el análisis metabolómico son la espectrometría de masas, que generalmente incluye un paso de separación cromatográfica, y la espectroscopia de RMN, dado que ambas plataformas proporcionan una amplia información estructural y conformacional sobre múltiples clases químicas en un procedimiento analítico único. Las dos técnicas poseen diferentes ventajas y desventajas desde el punto de vista técnico, y suelen ofrecer información complementaria (Lenz et al., 2007). Ambos métodos proporcionan información sobre un amplio conjunto de metabolitos, sin tener que preseleccionar qué analitos deben ser detectados. Esto nos permite diseñar experimentos basados en el razonamiento inductivo para generar nuevas hipótesis, siendo una vía potencial de descubrimiento de conocimiento a través del holismo. Además, los dos métodos pueden emplearse para identificar las estructuras de los metabolitos, y para medir las concentraciones relativas y absolutas.
OBJETIVOS DE LA METABOLÓMICA
Todos los estudios metabolómicos dan lugar a un conjunto de resultados complejo, que requiere un software específico para su visualización y métodos quimiométricos y bioinformáticos para su interpretación. El objetivo de estos experimentos es generalmente el de obtener huellas bioquímicas que puedan ser útiles desde un punto de vista diagnóstico o de clasificación, y, en un segundo lugar, el de identificar las sustancias que contribuyen a dicho diagnóstico o clasificación, ya que éstas conforman un conjunto complejo de biomarcadores que puede ayudar a definir el contexto biológico o clínico y explicar los mecanismos bioquímicos relacionados con los cambios observados, y así ampliar el conocimiento existente.
ESPECTROMETRÍA DE MASAS (EM O MS) Y RESONANCIA MAGNÉTICA NUCLEAR (RMN)
Tanto la EM como la RMN se emplean de forma rutinaria en los estudios metabolómicos.
La EM es una herramienta de análisis metabólomico muy poderosa, ya que provee una única mezcla de sensibilidad, rapidez, calidad y análisis potencialmente cuantitativo de una gran cantidad de metabolitos. Puede ser usada sola o en combinación con otras técnicas de cromatografía. Los análisis de EM requieren una preparación exhaustiva de la muestra y una extracción de la misma utilizando solventes orgánicos que pueden provocar la pérdida de algunos compuestos.
La RMN es una técnica no destructiva, no invasiva, y que no modifica el equilibrio biológico, y proporciona una información detallada sobre las estructuras moleculares en solución. Además la espectroscopia de RMN es una técnica muy fiable para las aplicaciones metabolómicas que requieren un alto grado de reproducibilidad.
Ambas técnicas permiten la detección simultánea de un amplio rango de metabolitos estructuralmente diversos y la detección en bajas concentraciones de los mismos.
Es importante tener en cuenta que, sea cual sea el origen de las muestras, deben evitarse fuentes potenciales de artefactos durante el proceso de recogida de las muestras biológicas, con el fin de obtener resultados que posean una buena calidad y fiabilidad.
Análisis estadístico multivariable
Los métodos de análisis estadístico multivariable proporcionan un medio de optimizar la extracción de la información contenida en los espectros de EM y RMN de mezclas complejas. Una vez realizados todos los pasos anteriores, lo que se tiene es una serie (uno por muestra) de perfiles multivariable que representan un punto en un espacio K-dimensional, y cuya posición (coordinadas) depende de los valores de cada uno de los descriptores (o metabolitos) que describen el sistema (la muestra). Cuando se trata de analizar una serie de perfiles, resulta muy útil la aplicación de lo que se denominan métodos basados en la proyección. Los métodos de proyección convierten tablas de datos multidimensionales en modelos de dimensionalidad. Esto permite detectar además agrupaciones y tendencias, ya que puntos que se encuentran cercanos corresponden a perfiles multivariables relativamente similares. De forma opuesta, puntos que se encuentran alejados unos de otros presentan descriptores muy diferentes.
Un ejemplo de lo explicado podemos verlo en la Figura 2, donde puede apreciarse la utilidad de esta herramienta para comprender los patrones que subyacen bajo la gran cantidad de información generada en los estudios metabolómicos. Quedan patentes las relaciones entre las distintas muestras, pudiendo describir agrupamientos, tendencias e influencias.
Aunque los métodos de proyección descritos forman la base de los estudios metabolómicos, los análisis estadísticos dependen del tipo de estudio que se esté desarrollando. Existe una gran variedad de métodos que pueden emplearse en el análisis de datos metabolómicos. La principal diferencia entre todos ellos es si se dispone o no de información previa sobre la clasificación de muestras en clases distintas, o si se sospecha la existencia de nuevas clases. Sin duda alguna, los métodos más empleados de análisis multivariable son los denominados PCA (análisis de componentes principales (Holmes, 1998)) y PLS (mínimos cuadrados parciales (Wold et al., 1984)). El PCA es un método no supervisado que se emplea para revelar la estructura interna de un conjunto de datos sin que medie ningún tipo de conocimiento previo del sistema. PLS, sin embargo, es un método supervisado para regresión. En este caso, el objetivo es predecir las respuestas en un conjunto de datos. Empleando una barrera de corte, PLS puede emplearse también para análisis basados en la clasificación y discriminación (PLS-DA, “Partial Least Squares-Discriminant Analysis”).
Identificación de metabolitos
Dependiendo del tipo de análisis que se esté desarrollando, los estudios metabolómicos pueden requerir la identificación de los metabolitos presentes en las muestras biológicas analizadas. Este objetivo puede alcanzarse mediante la comparación entre los desplazamientos químicos obtenidos en los espectros de EM o RMN y aquellos disponibles en bases de datos como la MMCD (Madison Metabolomics Consortium Database) (Cui et al., 2008)) o la HMDB (Human Metabolome DataBase) (Wishart et al., 2007). Alternativamente, existen programas (p.ej., “Analysis of MIXtures”, AMIX, Bruker BioSpin) que permiten realizar todas las etapas descritas anteriormente (selección de regiones de interés, segmentación del espectro, normalización, análisis estadístico) y que contienen módulos (B-Bioref-Code, Bruker BioSpin) que permiten la identificación y asignación de metabolitos presentes en mezclas complejas. Hay que tener en cuenta que las bibliotecas metabolómicas no son completas, y que no todos los metabolitos hallados tienen posibilidad de identificación.
ASESORAMIENTO METABOLÓMICO DE CALIDAD EMBRIONARIA
El metabolismo es intrínseco a la salud del embrión, por lo que muchas investigaciones se han concentrado en desarrollar marcadores metabólicos no invasivos de capacidad de desarrollo, como el consumo de oxígeno, reacciones REDOX, el consumo energético a través de Na+, K+ y ATPasa o el turnover de aminoácidos (Houghton et al., 2004).
Las últimas investigaciones en las técnicas moleculares están definiendo perfiles génicos, proteicos y metabolómicos para intentar detectar cuál es el ovocito o embrión más hábil, entendiendo esta habilidad como su capacidad para implantar (Katz-Jaffe et al., 2006; Patricio et al., 2007; Brison et al., 2007; Seli et al., 2007).
Actualmente se están desarrollando técnicas cuantitativas para el asesoramiento no invasivo del metabolismo embrionario, y es el objetivo de investigaciones actuales el determinar su valor como predictores de viabilidad embrionaria y embarazo (Seli et al., 2007).
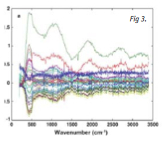
Seli y col. publicaron en 2007 el primer estudio con un enfoque metabolómico para estudiar la viabilidad del embrión humano. Ellos utilizaron como plataforma metabolómica de análisis el Near-infrared and Raman spectroscopy, para analizar y comparar el perfil metabolómico utilizando medios condicionados provenientes de embriones que implantaron frente a los que no implantaron. Aunque no encontraron marcadores específicos, si obtuvieron distintos espectros entre ambos grupos, siendo posible diferenciar fácilmente ambas poblaciones La Fig 3 muestra los distintos espectros que obtuvieron de los medios de cultivo embrionario analizados de cada una de las muestras utilizadas en el estudio (Seli et al., 2007).
El mismo grupo, en 2008, publica la identificación de marcadores de viabilidad utilizando análisis metabolómico mediante NMR, en un estudio retrospectivo realizado en 34 pacientes.
Explican encontrar diferencias significativas en concentraciones de glutamato entre embriones procedentes de embarazos y/o partos y embriones procedentes de fallo de implantación (Seli et al., 2008).
Otra aplicación de la metabolómica en el campo de la Reproducción Humana es el que estamos llevando en el Instituto Valenciano de Infertilidad, con el apoyo de la Fundación IVI. Nuestro objetivo es desarrollar un diagnóstico preimplantacional no invasivo con la ayuda de estas nuevas tecnologías. Pretendemos encontrar un perfil metabolómico embrionario que nos permita discriminar los embriones normales de los embriones aneuploides, utilizando el medio de cultivo donde éstos se desarrollan.
La instrumentación de análisis metabolómico que estamos utilizando es la Espectrometría de Masas asociada a UPLC. El sistema de UPLC ha sido diseñado para detectar partículas pequeñas (1,7 mm) y encontrar así perfiles de alta resolución. Por lo tanto, este sistema es óptimo para el análisis de muestras de alta complejidad, como nuestros medios de cultivo, debido al alto poder de separación metabólica y resolución de picos.
En los análisis llevados a cabo hasta la fecha, hemos observado que el perfil metabólico de los medios de cultivo de los embriones humanos parece discriminar entre embriones cromosómicamente normales y anormales, aunque existen limitaciones debidas a la baja cantidad de metabolitos presentes en las muestras.
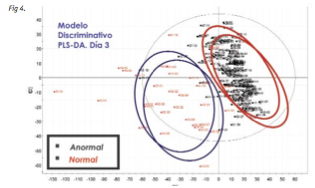
La Fig 4 es un score-plot donde cada punto representa un medio de cultivo embrionario, lo que nos permite apreciar la relación que hay entre las muestras. La tendencia observada es que muestras procedentes de embriones diagnosticados normales mediante diagnóstico preimplantacional para screening de aneuploidías (PGD-AS) se concentran en un área del diagrama de coordenadas, y muestras procedentes de embriones con diagnóstico de aneuploidías se concentran en un área distinta.
DISCUSIÓN
El método de evaluación tradicional embrionario tiene una capacidad limitada para seleccionar embriones con desarrollo potencial. Hay una clara necesidad de mejora en nuestro trabajo diario; deberíamos ser capaces de aumentar las tasas de gestación y disminuir el riesgo de gestación múltiple. Actualmente existen nuevas tecnologías en desarrollo para conseguir este objetivo, pudiendo ser tecnologías que utilicemos en un futuro como herramientas útiles de selección embrionaria, así como tecnologías que nos permitan expandir el conocimiento de la fisiología embrionaria con el objetivo de aumentar la eficacia de las Técnicas de Reproducción Asistida.
La capacidad de evaluar la producción y consumo metabolómico de un embrión individual nos llevaría a una mejor comprensión de la función celular y fisiología en etapas específicas de la embriogénesis. Por otra parte, este enfoque nos ayudaría a mejorar los medios de cultivo de embriones y podríamos identificar las interacciones entre los embriones y el epitelio uterino materno antes de la implantación.
Referencias
Beecher,CWW. The human metabolome. In metabolic profiling: its role in biomarker discovery and gene function analysis, 2003. pp. 311-319.
Brison DR, Hollywood K, Arnesen R, Goodacre R. Predicting human embryo viability: the road to non-invasive análisis of the secretome using metabolomic footprinting. Reproductive BioMedicine Online 2007; 15. 296-302.
Cui Q et al. Metabolite identification via the Madison Metabolomics Consortium Database. Nat Biotechnol 2008; 26: 162-164.
Dunn W, Ellis D. Metabolomics : current analytical platforms and methodologies. Trends in analytical chemistry 2005; 24: 285- 294.
Fiehn O. Metabolomics – the link between genotypes and phenotypes. Plant Mol Biol 2002; 48: 155-71.
Goodacre R, Vaidyanathan S, Dunn W, Harrigan G and Kell D. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends in Biotechnology 2004; 22: 245-252.
Holmes E, Nicholson J, Nicholls A, Lindon J, Connor S, Polley S, Connelly J. The identification of novel biomarkers of renal toxicity using automatic data reduction techniques and PCA of proton NMR spectra of urine Chemometrics and Intelligent Laboratory Systems. 1998; 44: 245-255.
Houghton F, Leese H. Metabolism and developmental competence of the preimplantational embryo. Europ Jour of Obst Gyn Reprod Bio 2004.115S. S92-S96.
Johnson HE, Broadhurst D, Goodacre R, Smith AR. Metabolic fingerprinting of salt-stressed tomatoes. Phytochemistry 2003; 62: 919-28.
Katz-Jaffe M, Gardner D, Schoolcraft W. Proteomic analysis of individual human embryos to identify novel biomarkers of development and viability. Fertility and Sterility 2006;1.
Kell D et al. (1999). Technological and medical implications of metabolic control analysis. Kluwr Academic Publishers, London, p3.
Lenz EM, Wilson ID. Analytical Strategies in Metabonomics. J Proteome Res 2007; 6: 443-458.
Nicholson JK, Lindon JC, Holmes E. ‘Metabonomics’: understanding the metabolic responses of living system to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data. Xenobiotica 1999; 29: 1181-9.
Oliver SG et al. Systematic functional analysis of de yeast genome. Trends biotechnol 1988; 16: 373-378
Patricio P, Fragouli E, Bianchi V, Borini A, Wells D. Molecular methods for selection of the ideal oocyte. Reproductive BioMedicine Online 2007; 15: 346-353.
Scott R, Seli E, Miller K, Sakkas D, Scott K, Burns D. Noninvasive metabolomic profiling of embryo culture media using Raman spectroscopy predicts embryomic reproductive potential: a prospective blinded pilot study. Fertyl Steril 2008; 90: 77-83.
Seli E, Sacas D, Scott C, Kwok S, Rosendahl S, Burns D. Non invasive metabolomis proflling of embryo culture media using Raman and near-infraread spectroscopy correlates with reproductive potencial of embryos in women undergoing in vitro fertilization.Fertyl Steril 2007; 88: 1350–7.
Seli E, Botros L, Sakkas D, and Burns DH. Noninvasive metabolomic profiling of embryo culture media using proton nuclear magnetic resonance correlates with reproductive potential of embryos in women undergoing in vitro fertilization. Fertil Steril 2008; 90: 2183-89.
Singh R, Sinclair K. Metabolomics: approache to assessing oocyte and embryo quality, Theriogenology 2007, doi:10.1016.
Urbanczyk-Wochniak E, Luedemann A, Kopka J, Selbig J, Roessner-Tunali U, Willmitzer L, Fernie AR. Parallel analysis of transcript and metabolic profile. EMBO Rep 2003; 4: 989- 992.
Waidyanathan S. et al. Explanatory optimisation of protein mass spectrometry via genetic search. Anal.Chem 2003; 75.
Whitfield PD, German A and Noble P. Metabolomics: and emerging post-genomic tool for nutrition. British Journal of Nutrition. 2004; 92: 549-555.
Wishart DS, et al. HMDB: the Human Metabolome Database. Nucleic Acids Res. 2007; 35. D521-D526.
Wold S, et al. The Collinearity Problem in Linear Regression. The Partial Least Squares (PLS) Approach to Generalized Inverses J Sci Stat Comput 1984; 5: 735-743.