INFLUENCIA DE LOS GENES DE EFECTO MATERNO Y TRANSICIÓN MATERNO-CIGÓTICA
Tania Moreno e Ismael Henarejos
tania.moreno1994@gmail.com
ihc.europa@gmail.com
La embriogénesis humana es un proceso que requiere de eventos bien orquestados y coordinados. Factores almacenados en el ovocito denominados genes de efecto materno (MEG, del inglés Maternally Expressed Genes) se expresarán tras la fecundación para el correcto desarrollo del embrión. Los MEG están involucrados en la remodelación de histonas, el control del huso mitótico, la formación del retículo endoplásmico (RE), la distribución de orgánulos, la activación de genes embrionarios y la impronta genómica, entre otros. En los últimos años, se han determinado varias proteínas codificadas por estos genes, las cuales son clave para la embriogénesis.
Tras una o varias rondas de escisión de blastómeros, el genoma del cigoto comienza a transcribirse y convirtiéndose en el responsable del desarrollo embrionario, mientras que los transcritos maternos se degradan gradualmente. Que esta transición materno-cigótica se lleve a cabo correctamente es una condición vital para la supervivencia del cigoto, pues problemas en este proceso pueden acarrear el bloqueo de los embriones y su degeneración. Sin embargo, son todavía desconocidos los genes implicados en este proceso, así como los marcadores de pluripotencia en el blastocisto y los mecanismos moleculares que subyacen a estos procesos.
En esta revisión se resumen los genes y transcritos implicados en la embriogénesis temprana y en la transición materno-cigótica. Estamos ante un campo de investigación que ha ganado interés en los últimos años debido a la búsqueda de biomarcadores que nos puedan dar información sobre la viabilidad y el potencial de desarrollo embrionario. Esto nos permitiría mejorar y modificar los tratamientos de reproducción asistida (TRA).
INTRODUCCIÓN
Durante el crecimiento del ovocito se van acumulando transcritos de los MEG cuya traducción se encuentra inhibida. Unos transcritos serán traducidos para propiciar el bloqueo de la polispermia y otros serán transcritos tras la fecundación por el espermatozoide, ya que están implicados en diversos procesos a partir de este momento (Kim and Lee, 2014). Ambos genomas, materno y paterno, son necesarios para el éxito del desarrollo. Sin embargo, los gametos tienen una carga desigual para garantizar el éxito en el desarrollo, desempeñando un papel de menor importancia en la fecundación y en la embriogénesis temprana los componentes celulares almacenados en el espermatozoide (Bruce, 2013).
Experimentos con microarrays y análisis proteómicos han definido los patrones globales de expresión génica en el desarrollo temprano y proporcionan información sobre el papel de los genes específicos durante los primeros estadios del desarrollo embrionario. Los estudios genéticos han demostrado que los MEGs afectan a múltiples procesos, incluyendo la formación pronuclear y la fusión (Philipps et al., 2008; Wu et al., 2003), la primera división celular (Burns et al., 2003; Tang et al.,2007), la transcripción de genes embrionarios (Bultman et al.,2006; Ramos et al., 2004) y la embriogénesis (Li et al., 2008a; Ma et al., 2006; Payer et al., 2003; Roest et al., 2004; Tong et al., 2000).
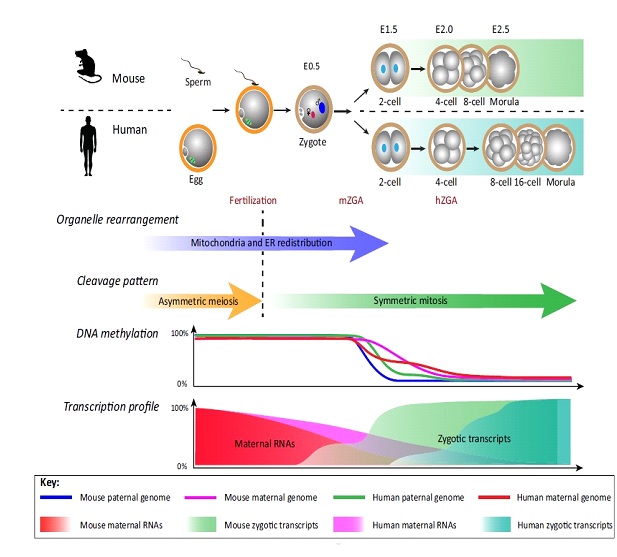
Durante las primeras divisiones embrionarias, los transcritos de origen materno irán desapareciendo gradualmente, comenzando así la transición materno-cigótica (ilustración 1). Este evento de degradación del 90% de los transcritos maternos, ocurre fundamentalmente en el estadio de 2 células en ratones, en el estadio de 4 células en humanos y en el estadio de 8 a 16 células en ganado (Bruce, 2013). El resultado de esta transición es la expresión de los genes del embrión. El pico de máxima expresión ocurre en la etapa de 8 células, lo cual señaliza la activación del genoma del cigoto (ZGA, del inglés zygotic genome activation).
La transición materno-cigótica es esencial para el desarrollo embrionario ya que coordina la división celular y la activación de genes del cigoto con el objetivo de preparar al embrión para la diferenciación celular y el posterior desarrollo, proveyendo de los sustratos moleculares necesarios para iniciar la gastrulación y especificación en determinadas capas y líneas celulares (endodermo, ectodermo y mesodermo) (Jukam et al., 2017).
En esta revisión expondremos en qué procesos a día de hoy se conoce la participación de los MEG, así como los últimos avances acerca de los procesos relacionados con los perfiles de expresión génica durante el desarrollo embrionario humano preimplantacional, la transición mateterno-cigótica, la ZGA y las conclusiones que se pueden extraer de los transcriptomas de estas fases tempranas del desarrollo embrionario.
Ilustración 1. Transición del ovocito al embrión temprano en el ratón y en el ser humanos. En los mamíferos, esta transición comienza con la fecundación del ovocito por parte del espermatozoide, lo que da inicio a una serie de cambios; a nivel celular se pasa de unas divisiones asimétricas propias de la meiosis a divisiones simétricas mitóticas; a nivel de orgánulos, se redistribuyen dinámicamente las mitocondrias y el retículo endoplasmático; a nivel molecular, los genomas de la madre y del embrión son desmetilados en patrones activos y pasivos; los transcritos de la madre son degradados gradualmente tras la fecundación mientras que el genoma del embrión es activado tras los primeros ciclos celulares. En el ratón, la activación genómica ocurre en la fase de dos células, mientras que en el humano esto ocurre en la transición de 4 a 6 células (Lu et al., 2017).
DISCUSIÓN
GENES Y TRANSCRITOS DEL OVOCITO
Tras la fecundación, los MEG se encargan del procesamiento del genoma masculino, cuya participación es necesaria para la embriogénesis. A continuación, eliminan los RNA y proteínas maternas y seguidamente activan el genoma embrionario, lo cual es esencial para el desarrollo embrionario más allá del tercer ciclo celular.
Durante el crecimiento de los ovocitos de ratón, el diámetro de estos aumenta de ~10 µm a 80 µm. El genoma materno se transcribe y estos transcritos se acumulan (~100 pg) hasta que los ovocitos alcanzan un diámetro de ~65 µm. La mayoría de los transcritos se traducen directamente en proteínas, muchas de las cuales han sido catalogadas. Sin embargo, otros transcritos permanecen en estado latente, y se activan más adelante en la ovogénesis mediante un proceso de poliadenilación.
A continuación, se describen los principales procesos desde la formación del embrión hasta su desarrollo a blastocisto en los que están implicados los MEG:
Bloqueo de la poliespermia
El bloqueo de la poliespermia es de vital importancia en la fecundación ya que la fecundación por más de un espermatozoide podría acarrear problemas de aneuploidías y fallos en el desarrollo embrionario. Uno de los genes implicadas es Uchl1, el cual, codifica para una proteína de fusión en la membrana ovocitaria (Kim and Lee, 2014).
Remodelación de histonas
La producción eficiente de histonas para el empaquetamiento del DNA recién replicado es particularmente importante para la división celular adecuada y el control epigenético durante las etapas iniciales en la implantación.
El factor nuclear de histona P (Hinfp) es el único factor de transcripción conocido para la expresión del gen de la histona H4, el cual se une directamente a un elemento específico del promotor de H4 para regular la transcripción. La deleción de Hinfp en fibroblastos embrionarios genera inestabilidad genómica y compromete la viabilidad celular. Esto explicaría el espaciamiento, el estrés replicativo, la alteración en la síntesis de DNA y la aneuploidía (Ghule et al., 2014). Los mRNA de histonas no se pasan a las células hijas en la mitosis del embrión porque se degradan rápidamente al completar la fase S. Esta degradación selectiva de los mRNA de histona provoca que se requiera de una síntesis de novo durante cada ciclo de división. Por lo tanto, una pregunta clave es cuándo y cómo las células activan la expresión de Hinfp (Ghule et al., 2016). Mediante knockout, se observó que Hinfp produce transcritos activamente durante la ovogénesis y su expresión cigótica protege a los embriones de la deficiencia de histonas (Ghule et al., 2016).
Por otro lado, el gen Brm. cuya subunidad catalítica Brg1 es un factor de supervivencia celular. Es esencial para la ZGA y está vinculado también a la modificación de histonas, en particular de la histona H3-K4 (Kim and Lee, 2014). Otro factor transcripcional, Ctcf, se ha propuesto como candidato por su función MEG ya que su déficit afecta a la ZGA en etapas tempranas al reducir la metilación de la histona H19 y la disminución por tanto en la competencia del desarrollo embrionario (Kim and Lee, 2014).
El complejo materno subcortical (SCMC)
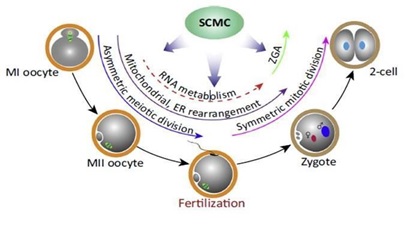
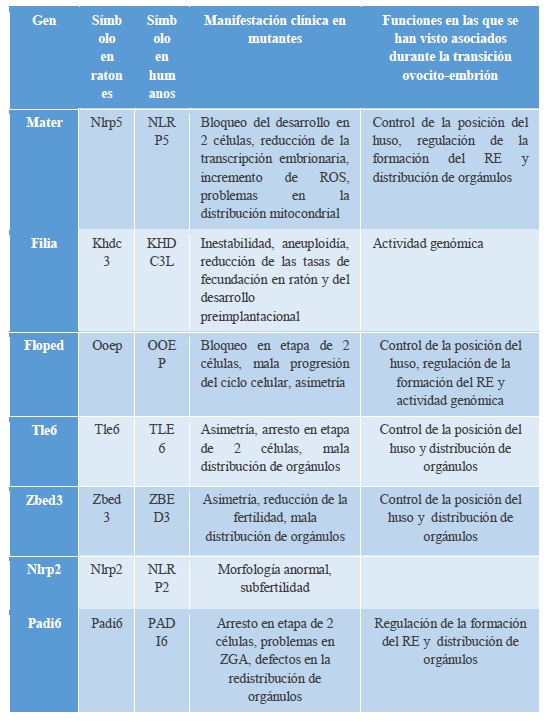
Se ha estudiado el papel de un complejo funcional de origen materno y almacenado en el ovocito de ratón, el SCMC, como posible modulador de varios de los procesos necesarios la transición materno-cigótica (Lu et al., 2017). Este complejo integra múltiples proteínas codificadas por los MEG y aparentemente su función se encuentra conservada en los mamíferos. Comprende Mater, Pillia, Floped, Tle6, Zbed3, Nlrp2 y posiblemente Padl6 (Tabla I). Se determinó que todas las proteínas que forman parte del complejo SCMC en el ratón presentan homólogos en el humano y se encuentran conservados para un gran porcentaje de sus dominios. Esto lleva a la idea de que la exploración de la función de SCMC en modelos de ratón podría conllevar a planteamientos terapéuticos para las causas de infertilidad humana y abortos espontáneos, además de la posibilidad de que un SCMC humana podría existir (hSCMC), pero son necesarios más estudios al respecto (Li et al., 2013, Lu et al., 2017).
Este complejo está asociado a muchas funciones en la transición de ovocito a embrión (Ilustracion 2):
Controla la posición del huso por la regulación de actina del citoesqueleto: La transición de ovocito a embrión va acompañada del cambio de una meiosis asimétrica a una mitosis simétrica, posiblemente bajo el control de la F-actina. Se ha observado que la mala posición del huso está asociada a mutantes de Tle6, Mater, Fliped y Zbed3 (Lu et al., 2017).
Regulan la formación del retículo endoplasmático (RE): El retículo endoplasmático se distribuye por todo el citoplasma de los ovocitos. Se ha especulado que puede servir como almacenamiento de RNA y ribosomas maternos. Varios estudios recientes han demostrado que la función de éste está relacionada con SCMC puesto que Floped, Mater y Padi6 residen en el RE (Lu et al., 2017).
Distribución de los orgánulos: Un estudio reveló que aquellos ovocitos donde no se expresa Mater exhibían un aumento de la actividad de sus mitocondrias, lo que condujo a un aumento en los niveles de las especies reactivas de oxígeno (ROS) pudiendo afectar a muchos eventos de señalización como, por ejemplo, la redistribución de RE que conlleva una disminución de las reservas de Ca2+ intracelular, provocando una detención del desarrollo. Esto se observó posteriormente también en mutantes Tle6, Padi6 y Zbed3. (Lu et al., 2017).
Linaje Celular: EL SCMC también ejercería un papel importante en el establecimiento del destino celular. Según lo observado en embriones de ratón, la disminución del contacto celular, junto con la exclusión de SCMC predice a la participación de este en el establecimiento del linaje celular. Aquellos embriones carentes de SCMC sólo eran capaces de convertir las células externas en ICM, mientras que las que sí lo contienen forman preferentemente el trofoectodermo. Que algunas células internas se conviertan en células del trofoectodermo podría reflejar la restauración del SCMC en ausencia del contacto célula a célula. La localización de Cdx2 y Floped uniéndose a homopolímeros de ribonucleótidos son ideas firmes de que el complejo tiene un papel en el secuestro de RNA en las células externas para influir en vías posteriores para el linaje. Por lo tanto, este complejo podría participar en el mantenimiento de la pluripotencia de las blastómeras (Li et al., 2010).
TRANSCRIPCIÓN MATERNO-CIGÓTICA
Ilustración 2. Efectos del SCMC en el ovocito y desarrollo embrionario (Lu et al., 2017)
Degradación de transcritos
La cantidad de mRNA disminuye en un 60% durante el primer ciclo celular y para la etapa de dos células, más del 90% del mRNA materno se ha degradado (Bachvarova, 1985; Paynton et al., 1988). Esta degradación gradual es debida a su desestabilización mediado por micro-RNA embrionario (Giraldez et al., 2006; Miccoli et al., 2017). Se postula la existencia de un conjunto de mecanismos para degradar de manera diferencial estos mRNA, entre los que destacan las secuencias 3ÚTR, Atg5 y Ago2 (Kim and Lee, 2014). Existen dos vías de degradación: la materna y la cigótica, siendo esta última dependiente de los transcritos cigóticos y se activa 2h después de la fecundación.
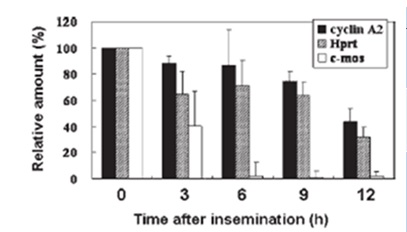
Algunos de los transcritos susceptibles a la degradación son proteínas que desempeñan papeles importantes durante la meiosis y que son perjudiciales para el desarrollo tras la fecundación. Destacan los genes H1oo, c-mos, tPA y gdf9 cuyas transcripciones se degradan rápidamente tras la fecundación. Esto se corrobora mediante RT-PCR donde se observó que ninguno de estos transcritos se expresaba durante el desarrollo embrionario preimplantacional (Gráfica 1) (Alizadeh et al., 2005).
Activación del genoma del cigoto
La ZGA en el embrión humano es un proceso que se considera gradual, comenzando a detectarse transcritos en el embrión de dos células (Vassena et al., 2011) y aumentando el número de estos transcritos a medida que crece el número de células y núcleos (B. Liu and Grosshans, 2017). Este modelo de activación se denomina “en ondas” y a día de hoy se sigue estudiando qué genes se expresan durante este proceso y cuál es su función (C. Liu et al., 2018, Miao et al., 2018). Pese a la importancia de este proceso, se desconocen exactamente los mecanismos moleculares que ocurren en esta fase del embrión humano.
Recientemente se ha realizado un estudio usando la técnica de edición genética CRISPR-Cas9 con el fin de sobreexpresar en mioblastos humanos la familia de genes DUX, la cual regula una gran cantidad de genes en la ZGA (De Iaco et al., 2017). La regulación génica que lleva a cabo la familia DUX ocurre cuando se unen a los promotores de los genes, resultando en un aumento de la transcripción. El estudio detectó mediante ARN-seq transcritos para estos factores de transcripción desde el ovocito hasta la fase de 4 células en el embrión. (De Iaco et al., 2017, Vassena et al., 2011). Los resultados del estudio sugieren que la familia DUX tendría un papel importante en actuar como factores pioneros del inicio de la transcripción del genoma del embrión humano, ya que permitirían el acceso de la maquinaria transcripcional a los sitios de inicio de la transcripción de estos genes (De Iaco et al., 2017)
Otro estudio destacable es el uso del CRISPR-Cas9 en embriones humanos para determinar el papel de OCT4/ POUF51 en la segmentación (Fogarty et al., 2017). OCT4 es uno de los marcadores de pluripotencialidad en el blastocisto, el cual si se silencia se observa que los embriones se bloquean en la segmentación, en concreto en la transición de 4 a 8 células. Por tanto, OCT4 asumiría un papel importante y previamente no detectado en la ZGA humana, y defectos en su expresión podrían comprometer la calidad y viabilidad de los embriones humanos. (Fogarty et al., 2017).
GENES Y TRANSCRITOS DEL EMBRIÓN
Transcriptómica del desarrollo embrionario humano durante el desarrollo preimplantacional y establecimiento del destino celular.
De manera general, unos 200 genes aumentan su expresión en la fase de 2 células, siendo genes relacionados con la biosíntesis y modificación de proteínas, regulados por factores maternos. Principalmente, se traducen proteínas de unión a ácido nucleico, proteínas ribosómicas e histonas, sugiriendo que la primera ola de síntesis de proteínas va dirigida a la formación de la maquinaria transcripcional y traduccional para posteriores fases del desarrollo (Vassena et al., 2011).
Aproximadamente unos 250 genes diferentes a los de los estadios anteriores aumentan su expresión al inicio de la fase de 4 células y en este momento se activa una maquinaria transcripcional reguladora cigótica. Este proceso se mantiene y crece de manera gradual y estable en fases posteriores de 8 y 10 células, donde continúa la biosíntesis de proteínas y la actividad transcripcional, alcanzando un pico de expresión en la etapa de 8 células (Vassena et al., 2011). Este pico de expresión se pierde en la transición a la mórula, donde se favorece la expresión de genes relacionados con el metabolismo de lípidos, aminoácidos, proteínas y carbohidratos, suceso que continua en la transición a blastocisto (Vassena et al., 2011). Los perfiles de expresión génica en el blastocisto se han estudiado más detenidamente, especialmente por su relación con las células madre embrionarias (Fassnacht and Ciosk, 2017, Stirparo et al., 2018). Además, la transcriptómica del blastocisto nos permite estudiar cómo se establece la pluripotencialidad a nivel génico, y qué genes se relacionan con la determinación del posible destino celular (ectodermo, mesodermo, endodermo) (D. V. Onichtchouk and Voronina, 2015).
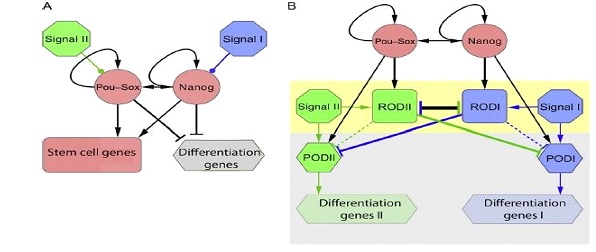
El establecimiento de la pluripotencialidad de manera general se debe a la regulación de 3 factores de transcripción: NANOG, OCT4/POUF51, y SOX2. Estos junto con el coactivador p300 regulan la expresión de genes uniéndose a potenciadores de la transcripción, en concreto, a secuencias específicas denominadas SOX-POU (D. V. Onichtchouk and Voronina, 2015) (Ilustración 3).
CONCLUSIÓN
La calidad del ovocito es un factor crítico que limita la eficacia de las TRA y el éxito del embarazo. Esto se debe a la participación de los factores maternos acumulados en el ovocito en diversos procesos del desarrollo embrionario temprano incluyendo bloqueo de la polispermia, remodelación de histonas, control del huso, formación del RE, distribución de orgánulo, activación de genes embrionarios, establecimiento de linaje y degradación de mRNA maternos. Sin embargo, a pesar de que diferentes estudios han demostrado la asociación entre perfiles de expresión génica y producción de embriones de calidad, la regulación de la calidad de los ovocitos sigue siendo poco conocida.
Comprender el transcriptoma del desarrollo embrionario humano es vital para interpretar los aspectos funcionales del genoma, sin embargo, las dificultades éticas y legales a la hora de realizar investigación con embriones humanos, han impedido que haya un gran número de estudios al respecto. Destacar que los estudios en ratones abren la posibilidad de nuevas aplicaciones en las TRA debido a la homología con los humanos.
AGRADECIMIENTOS
María José Escribá Pérez y Nuria Soler Balaguer por el apoyo y la ayuda en la redacción de este artículo.
Ilustración 3. Dos modelos posibles del establecimiento de la pluripotencia y mantenimiento de ésta. Las flechas con punta indican que la expresión es favorecida, mientras que las flechas con punta redonda indican represión de la expresión. La terminación en línea representa inhibición. (A) Modelo simplificado: En respuesta a unas condiciones ambientales, los genes OCT4/POUFSI, SOX2 y NANOG son activados y bajo un mecanismo de feedback positivo mantienen su propia expresión, estableciendo el estado de pluripotencia, caracterizado por la expresión de genes propios de células madres, mientras que los genes de diferenciación se encuentran inhibidos hasta que una serie de señales impliquen la transición a un estado diferenciado. (B) Modelo de transición en el embrión en el blastocisto tardío y en la gastrulación. Bajo el efecto del circuito POU·SOX·NANOG, se encuentran activados una serie de factores de represión de la diferenciación, RODI y RODII. Previo a la diferenciación existe un equilibrio entre las señales de diferenciación y la expresión de los ROD. En respuesta a unas señales específicas de tejido, aumentan su expresión, el equilibrio se rompe y un ROD de una línea celular determinada continua activo, mientras que la otra se inhibe, permitiendo la expresión de unos promotores de la diferenciación POD que transcribirán los genes relacionados con una línea celular determinada (D. Onichtchouk and Driever, 2016; D. V. Onichtchouk and Voronina, 2015; M. Zhang et al., 2017)
Gráfica 1. Degradación de varios mRNA. Se seleccionaron C-mos, ciclina A2 y Hprt puesto que se expresan abundantemente en ovocitos. C-mos es una transcripción específica de ovocitos y no se detecta en embriones de una sola célula, mientras que las ciclinas A2 y Hprt se detectan en ambos, ovocitos y embriones tempranos y las cantidades de estas transcripciones no difieren notablemente en embriones de una y dos células (Alizadeh et al., 2005).
Tabla 1. Componentes del SCMC.
Referencias
Alizadeh Z, Kageyama S, Aoki F. Degradation of Maternal mRNA in Mouse Embryos: Selective Degradation of Specific mRNAs After Fertilization. 2005; 281-290.
Bruce AW. Generating different genetic expression patterns in the early embryo: Insights from the mouse model. 2013:6; 586-592.
Bultman SJ, Gebuhr TC, Pan H, Svoboda P, Schultz RM, Magnuson T. Maternal BRG1 regulates zygotic genome activation in the mouse. Genes Dev 2006:13; 1744-1754.
Burns KH, Viveiros MM, Ren Y, Wang P, DeMayo FJ, Frail DE, et al. Roles of NPM2 in Chromatin and Nucleolar Organization in Oocytes and Embryos. Science 2003:5619; 633 LP-636.
Denomme MM, Mann MRW. Maternal control of genomic imprint maintenance. 2013:6; 629- 636.
De Iaco A, Planet E, Coluccio A, Verp S, Duc J, Trono D. DUX- family transcription factors regulate zygotic genome activation in placental mammals. Nat Genet 2017;49:941- 945.
Fassnacht C, Ciosk R. Cell Fate Maintenance and Reprogramming During the Oocyte-to- Embryo Transition. Results Probl Cell Differ 2017;59:269-286.
Fogarty NME, McCarthy A, Snijders KE, Powell BE, Kubikova N, Blakeley P, et al. Genome editing reveals a role for OCT4 in human embryogenesis. Nature 2017;550:67-73.
Ghule PN, Xie R-, Medina R, Colby JL, Jones SN, Lian JB, et al. Fidelity of histone gene regulation is obligatory for genome replication and stability. Mol Cell Biol 2014:14; 2650-2659.
Ghule PN, Xie R, Colby JL, Rivera-pérez J,A., Jones SN, Lian JB, et al. Maternal expression and early induction of histone gene transcription factor Hinfp sustains development in pre-implantation embryos. Dev Biol 2016:2; 311-320.
Giraldez AJ, Mishima Y, Rihel J, Grocock RJ, Van Dongen S, Inoue K, et al. Zebrafish MiR- 430 Promotes Deadenylation and Clearance of Maternal mRNAs. Science 2006:5770; 75 LP-79.
Jukam D, Shariati SAM, Skotheim JM. Zygotic Genome Activation in Vertebrates. Dev Cell 2017;42:316-332.
Kim K, Lee K. Maternal effect genes: Findings and effects on mouse embryo development embryonic arrest. 2014:2; 47- 61.
Liu B, Grosshans J. Link of Zygotic Genome Activation and Cell Cycle Control. Methods Mol Biol 2017;1605:11-30.
Liu C, Ma Y, Shang Y, Huo R, Li W. Post-translational regulation of the maternal-to-zygotic transition. Cell Mol Life Sci 2018;.
Li L, Baibakov B, Dean J. A Subcortical Maternal Complex Essential for Pre-implantation Mouse Embryogenesis. 2008:3; 416-425.
Li L, Lu X, Dean J. The maternal to zygotic transition in mammals. Mol Aspects Med 2013:5; 919- 938.
Li L, Zheng P, Dean J. Maternal control of early mouse development. 2010:; 859-870.
Li X, Ito M, Zhou F, Youngson N, Zuo X, Leder P, Ferguson-Smith A. A maternal-zygotic effect gene Zfp57 maintains both maternal and paternal imprints. 2008:4; 547-557.
Lu X, Gao Z, Qin D, Li L. A Maternal Functional Module in the Mammalian Oocyte-To- Embryo Transition. Trends Mol Med 2017:11; 1014-1023.
Ma J, Zeng F, Schultz RM, Tseng H. Basonuclin: a novel mammalian maternal-effect gene. Development 2006:10; 2053 LP-2062.
Miao YL, Gambini A, Zhang Y, Padilla- Banks E, Jefferson WN, Bernhardt ML, et al. Mediator complex component MED13 regulates zygotic genome activation and is required for postimplantation development in the mouse. Biol Reprod 2018;.
Miccoli A, Dalla L, Carnevali O. The maternal control in the embryonic development of zebrafish. Gen Comp Endocrinol 2017:; 55- 68.
Onichtchouk D, Driever W. Zygotic Genome Activators, Developmental Timing, and Pluripotency. Curr Top Dev Biol 2016;116:273-297.
Onichtchouk DV, Voronina AS. Regulation of Zygotic Genome and Cellular Pluripotency. Biochemistry (Mosc) 2015;80:1723- 1733.
Payer B, Saitou M, Barton SC, Thresher R, Dixon JPC, Zahn D, et al. Stella is a maternal effect gene required for normal early development in mice. 2003:23; 2110-2117.
Paynton BV, Rempel R, Bachvarova R. Changes in state of adenylation and time course of degradation of maternal mRNAs during oocyte maturation and early embryonic development in the mouse. Dev Biol 1988:2; 304-314.
Philipps DL, Wigglesworth K, Hartford SA, Sun F, Pattabiraman S, Schimenti K, et al. The dual bromodomain and WD repeat-containing mouse protein BRWD1 is required for normal spermiogenesis and the oocyte-embryo transition. Dev Biol 2008:1; 72-82.
Ramos SBV, Stumpo DJ, Kennington EA, Phillips RS, Bock CB, Ribeiro-Neto F, et al. The CCCH tandem zinc-finger protein Zfp36l2 is crucial for female fertility and early embryonic development. Development 2004:19; 4883 LP-4893.
Schulz KN, Bondra ER, Moshe A, Villalta JE, Lieb JD, Kaplan T, et al. Zelda is differentially required for chromatin accessibility, transcription factor binding, and gene expression in the early Drosophila embryo. Genome Res 2015;25:1715-1726.
Stirparo GG, Boroviak T, Guo G, Nichols J, Smith A, Bertone P. Integrated analysis of single-cell embryo data yields a unified transcriptome signature for the human pre-implantation epiblast. Development 2018;145:10.1242/dev.158501.
Tang F, Kaneda M, O. Carroll D, Hajkova P, Barton SC, Sun YA, et al. Maternal microRNAs are essential for mouse zygotic development. Genes Dev 2007:6; 644-648.
Tong ZB, G., and Nelson LM. Mater, a maternal effect gene required for early embryonic development in mice. 2000:; 267-269
Vassena R, Boue S, Gonzalez-Roca E, Aran B, Auer H, Veiga A, Izpisua Belmonte JC. Waves of early transcriptional activation and pluripotency program initiation during human preimplantation development. Development 2011;138:3699-3709.
Wu X, Viveiros MM, Eppig JJ, Bai Y, Fitzpatrick SL, Matzuk MM. Zygote arrest 1 (Zar1) is a novel maternal-effect gene critical for the oocyte-to-embryo transition. 2003:; 187- 191.